欢迎关注”生信修炼手册”!
meta-analysis对多个独立研究的成果进行综合评估,该技术在医学,心理学等领域早已广泛使用。虽然该技术的理论基础早已成熟,但是在GWAS分析领域,还是有很多困难需要去克服
GWAS分析SNP位点的数量很大,特别是基因型填充技术的出现,SNP位点数量达到了百万,千万的规模,对于软件而言,运行速度和内存消耗也是巨大挑战
不同study结果的存储格式不尽相同,需要统一的格式转换,特别是链的方向问题,需要调整成一个统一的ref allele
GWAS分析结果的准确性问题,群体分层等因素都需要被校正
为了有效的进行分析,我们需要使用成熟软件,本文要介绍的GWAMA就是一款很不错的软件,其运行效率特别高,适合针对大数量的SNP位点进行分析,对应的文章发表在BMC Bioinformatics上,链接如下
https://bmcbioinformatics.biomedcentral.com/track/pdf/10.1186/1471-2105-11-288
官方网址如下
https://www.geenivaramu.ee/en/tools/gwama
安装过程如下

采用C++进行开发,下载源代码解压,编译即可。官网也提供了配套的测试数据,可以用于测试该软件
该软件的基本用法如下
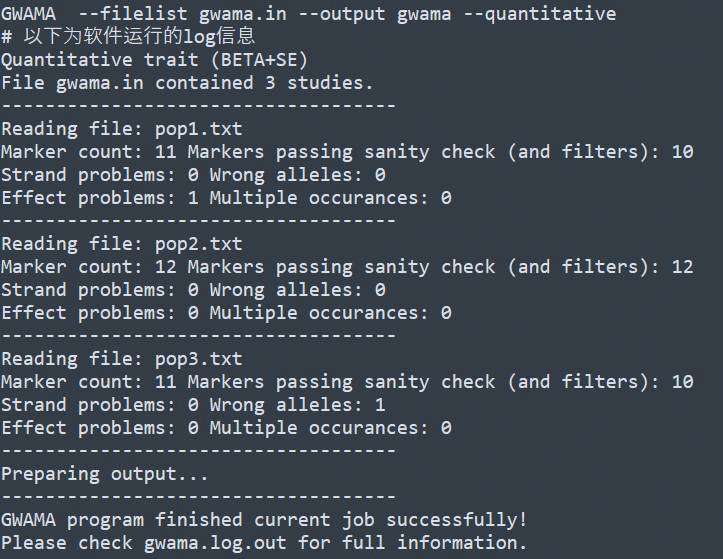
只需要一个输入文件,默认文件名称为gwama.in, 内容示意如下

每一行代表一个study的gwas分析结果,对应的文件格式如下
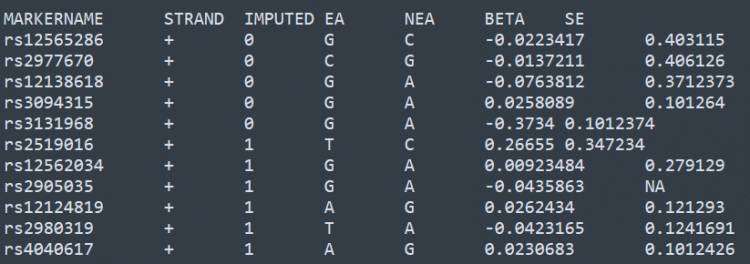
MARKERNAME代表SNP位点的名称,STRAND代表链的方向,IMPUTED代表该位点是否是填充得到的,EA代表tested allele, NEA代表other allele, 对于连续型的性状,要求输入BETA和SE两列,对于二分类的性状,要求输入OR, OR_95L, OR_95U3列,代表odd ratio以及95%置信区间。
默认输出结果保存在gwama.out文件中,列数较多,为了方便展示,我这里截取了部分并进行了转置,内容如下
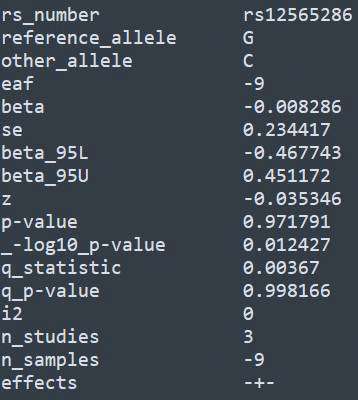
另外该软件还支持针对性别进行校正,更多用法请查看官方文档。
·end·
—如果喜欢,快分享给你的朋友们吧—
往期精彩
自己动手进行逻辑回归,你也可以!
odd ratio置信区间的计算,你学会了吗?
多元回归分析存在多重共线性了怎么办?
基因型与表型的交互作用如何分析,多元回归来搞定
曼哈顿图就够了吗?你还需要LocusZoom
GWAS做完了,下一步做什么?
逻辑回归or线性回归,傻傻分不清楚
没想到你是这个样子的置信区间
线性回归的这些细节,你都搞明白了吗?
3分钟掌握曼哈顿图的绘制
一文搞懂Q-Q plot图的含义
Cochran-Mantel-Haenszel检验在关联分析中的应用
plink中case/control关联分析细节解析
odd ratio值在关联分析中的含义
Cochran-Armitage趋势检验在关联分析中的应用
基因型填充
GWAS中的genotype imputation简介
基因型填充中的phasing究竟是什么
基因型填充前的质控条件简介
使用shapeit进行单倍型分析
gtool:操作genotype data的利器
使用IMPUTE2进行基因型填充
使用Beagle进行基因型填充
使用Minimac进行基因型填充
使用Eagle2进行单倍型分析
X染色体的基因型填充
文献解读|不同基因型填充软件性能的比较
Haplotype Reference Consortium:最大规模的单倍型数据库
Michigan Imputation Server:基因型填充的在线工具
CNV分析
aCGH芯片简介
aCGH芯片分析简介
基于SNP芯片进行CNV分析中的基本知识点
PennCNV:利用SNP芯片检测CNV
DGV:人类基因组结构变异数据库
dbvar:染色体结构变异数据库
DGVa:染色体结构变异数据库
CNVD:疾病相关的CNV数据库
DECIPHER:疾病相关的CNV数据库
全基因组数据CNV分析简介
使用CNVnator进行CNV检测
使用lumpy进行CNV检测
CNVnator原理简介
WES的CNV分析简介
XHMM分析原理简介
使用conifer进行WES的CNV分析
使用EXCAVATOR2检测WES的CNV
靶向测序的CNV分析简介
使用CNVkit进行CNV分析
DECoN:最高分辨率的CNV检测工具
TCGA
TCGA数据库简介
使用GDC在线查看TCGA数据
使用gdc-client批量下载TCGA数据
一文搞懂TCGA中的分析结果如何来
通过GDC Legacy Archive下载TCGA原始数据
使用GDC API查看和下载TCGA的数据
使用GDC下载TCGA肿瘤患者的临床信息
使用TCGAbiolinks下载TCGA的数据
使用TCGAbiolinks进行生存分析
使用TCGAbiolinks分析TCGA中的表达谱数据
使用TCGAbiolinks进行甲基化和转录组数据的联合分析
Broad GDAC:TCGA数据分析中心
使用cBioPortal查看TCGA肿瘤数据
UCSC Xena:癌症基因组学数据分析平台
GEPIA:TCGA和GTEx表达谱数据分析平台
TANRIC:肿瘤相关lncRNA数据库
SurvNet:基于网络的肿瘤biomarker基因查找算法
TCPA:肿瘤RPPA蛋白芯片数据中心
TCGA Copy Number Portal:肿瘤拷贝数变异数据中心
生存分析
生存分析详细解读
用R语言进行KM生存分析
使用OncoLnc进行TCGA生存分析
用R语言进行Cox回归生存分析
使用kmplot在线进行生存分析
肿瘤数据库
ICGC:国际肿瘤基因组协会简介
HPA:人类蛋白图谱数据库
Oncomine:肿瘤芯片数据库
ONGene:基于文献检索的肿瘤基因数据库
oncomirdb:肿瘤相关的miRNA数据库
TSGene:肿瘤抑癌基因数据库
NCG:肿瘤驱动基因数据库
mutagene:肿瘤突变频谱数据库
CCLE:肿瘤细胞系百科全书
mSignatureDB:肿瘤突变特征数据库
GTEx:基因型和基因表达量关联数据库
肿瘤免疫和新抗原
Cancer-Immunity Cycle:肿瘤免疫循环简介
TMB:肿瘤突变负荷简介
肿瘤微环境:Tumor microenvironment (TME)简介
肿瘤浸润免疫细胞量化分析简介
使用EPIC预测肿瘤微环境中免疫细胞构成
TIMER:肿瘤浸润免疫细胞分析的综合网站
quanTIseq:肿瘤浸润免疫细胞定量分析
The Cancer Immunome Atlas:肿瘤免疫图谱数据库
肿瘤新抗原简介
TSNAdb:肿瘤新抗原数据库
使用NetMHCpan进行肿瘤新抗原预测分析
Hi-C数据分析
chromosome-territories:染色质疆域简介
chromosome conformation capture:染色质构象捕获技术
3C的衍生技术简介
解密Hi-C数据分析中的分辨率
A/B compartment:染色质区室简介
TAD:拓扑关联结构域简介
chromatin loops:染色质环简介
Promoter Capture Hi-C:研究启动子区染色质互作的利器
使用HiCUP进行Hi-C数据预处理
Juicer:Hi-C数据处理分析的利器
Juicer软件的安装详解
Juicebox:Hi-C数据可视化利器
Juicer实战详解
HiC-Pro:灵活的Hi-C数据处理软件
HiC-Pro实战详解
3D Genome Browser:Hi-C数据可视化工具
HiCPlotter:Hi-C数据可视化工具
3CDB:基于3C技术的染色质互作信息数据库
3DIV:染色质空间互作数据库
4DGenome:染色质相互作用数据库
4D nucleome project:染色质三维结构研究必不可少的参考项目
3dsnp:SNP在染色质环介导的调控网络中的分布数据库
iRegNet3D:疾病相关SNP位点在三维调控网络中的作用
使用WashU Epigenome Browser可视化hi-c数据
HiGlass:高度定制的Hi-C数据可视化应用
Hi-C Data Browser:Hi-C数据浏览器
使用FitHiC评估染色质交互作用的显著性
使用TADbit识别拓扑关联结构域
使用pyGenomeTracks可视化hi-c数据
hi-c辅助基因组组装简介
文献解读|使用hi-C数据辅助埃及伊蚊基因组的组装
chip_seq数据分析
Chip-seq简介
chip_seq质量评估之计算样本间的相关性
chip_seq质量评估之查看抗体富集效果
chip_seq质量评估之PCA分析
chip_seq质量评估之coverage分析
chip_seq质量评估之FRiP Score
chip_seq质量评估之cross correlation
chip_seq质量评估之文库复杂度
depth, bedgraph, bigwig之间的联系与区别
bigwig归一化方式详解
使用igvtools可视化测序深度分布
使用UCSC基因组浏览器可视化测序深度分布数据
使用deeptools查看reads分布特征
使用phantompeakqualtools进行cross correlation分析
blacklist regions:NGS测序数据中的黑名单
MACS:使用最广泛的peak calling软件之一
MACS2 peak calling实战
使用SICER进行peak calling
使用HOMER进行peak calling
peak注释信息揭秘
PAVIS:对peak区域进行基因注释的在线工具
使用UPORA对peak进行注释
使用GREAT对peak进行功能注释
annoPeakR:一个peak注释的在线工具
使用ChIPpeakAnno进行peak注释
使用ChIPseeker进行peak注释
使用PeakAnalyzer进行peak注释
使用homer进行peak注释
利用bedtools预测chip_seq数据的靶基因
motif
关于motif你需要知道的事
详解motif的PFM矩阵
详解motif的PWM矩阵
使用WebLogo可视化motif
使用seqLogo可视化motif
使用ggseqlogo可视化motif
MEME:motif分析的综合性工具
使用MEME挖掘序列中的de novo motif
使用DREME挖掘序列中的de novo motif
使用MEME-ChIP挖掘序列中的de novo motif
chip_seq数据库
ENCODE project项目简介
FactorBook:人和小鼠转录因子chip_seq数据库
ReMap:人类Chip-seq数据大全
IHEC:国际人类表观基因组学联盟
Epifactors:表观因子数据库
GTRD:最全面的人和小鼠转录因子chip_seq数据库
ChIP-Atlas:基于公共chip_seq数据进行分析挖掘
Cistrome DB:人和小鼠的chip_seq数据库
chipBase:转录因子调控网络数据
unibind:human转录因子结合位点数据库
chip_seq在增强子研究中的应用
DENdb:human增强子数据库
VISTA:人和小鼠的增强子数据库
EnhancerAtlas:人和小鼠的增强子数据库
FANTOM5:人类增强子数据库
TiED:人类组织特异性增强子数据库
HEDD:增强子疾病相关数据库
HACER:human增强子数据库
SEdb:超级增强子数据库简介
dbSUPER:人和小鼠中的超级增强子数据库
dbCoRC:核心转录因子数据库
使用ROSE鉴定超级增强子
18年文章目录
扫描下方二维码,关注我们,解锁更多精彩内容!

生物信息入门
只差这一个
公众号